アルデヒドデヒドロゲナーゼ2の遺伝子型決定
ISOHAIR実験例概要
アルデヒドデヒドロゲナーゼ2遺伝子(ALDH2)について
アルデヒドデヒドロゲナーゼ2(ALDH2)は、飲酒後エタノールが代謝されてできるアルデヒドを酸化して代謝する酵素です。ALDH2の欠損はALDH2遺伝子の点突然変異によるものと考えられており、対立遺伝子の組み合わせから、正常型ホモ接合体(NN型)、ヘテロ接合体(MN型)、変異型ホモ接合体(MM型)の3種類の遺伝子型が知られています。
3種類のうちのどの遺伝子型を持っているかを調べることで、酒に強いか弱いかを判断することができます。正常型ホモ接合体は酒が飲めるタイプ、ヘテロ接合体は飲む事はできるがすぐ顔に出るタイプ、変異型ホモ接合体は全く飲めないタイプです。ALDH2欠損者においては、飲酒後、血中アセトアルデヒド濃度の上昇により、フラッシング反応(顔面紅潮、動悸、悪心、低血圧等)が見られます。
<NN 型> 正常型ホモ接合体 |
お酒が飲めるタイプ |
|---|---|
<MN 型> ヘテロ接合体 |
飲むことはできるがすぐ顔にでるタイプ |
<MM 型> 変異型ホモ接合体 |
全く飲めないタイプ |
▲このページのトップへ
実験例
3検体のヒトの毛髪からDNAを抽出し、正常型特異的増幅と変異型特異的増幅を行い、アルデヒドデヒドロゲナーゼ2遺伝子の遺伝子型を決定した。
実験条件
- 鋳型
ISOHAIRを用いてヒト毛髪より抽出したDNA - プライマー配列
・Forward : 5'-CAAATTACAGGGTCAACTGCT-3'
・Reverse-N(野生型): 5'-CCACACTCACAGTTTTCTCTTC-3'
・Reverse-M(変異型): 5'-CCACACTCACAGTTTTCTCTTT-3' - PCR用試薬
・Gene Taq NT(構成品: Gene Taq NT, 10 x Gene Taq Universal Buffer, 2.5mM each dNTP Mixture)
・d.d.H2O
鋳型 |
1 µl |
10 x Gene Taq Universal Buffer |
5 µl |
dNTP Mixture |
4 µl |
プライマー(Forward, 20 pmol/µl) |
1 µl |
プライマー(Reverse-NまたはM, 20 pmol/µl) |
1 µl |
d.d.H2O |
37.75 µl |
Gene Taq NT |
0.25 µl |
Total |
50 μl |
![]()
| 98°C | 1分 | - |
| 98°C | 20秒 | 35サイクル |
| 60°C | 20秒 | |
| 72°C | 45秒 | |
| 72°C | 5分 | - |
結果
Sample1ではReverse-Nのみ、Sample2ではReverse-N、Mの両方、Sample3ではReverse-Mのみバンドが確認できました。この結果から、Sample1がNN型(野生型ホモ)、Sample2がMN型(ヘテロ)、Sample3がMM型(変異型ホモ)であることが分かりました。
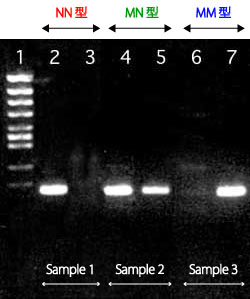 |
Lane1: Marker 5(φX174/Hinc II digest) Lane2: Sample1/Reverse-N(野生型) Lane3: Sample1/Reverse-M(変異型) Lane4: Sample2/Reverse-N(野生型) Lane5: Sample2/Reverse-M(変異型) Lane6: Sample3/Reverse-N(野生型) Lane7: Sample3/Reverse-M(変異型) 3% Agarose 21 |
▲このページのトップへ
補足情報
補足情報.1 プライマー配列について
上記実験例で紹介しているプライマー配列の設計は、下記論文を参考にしました。
"Characterization of the three genotypes of low Km aldehyde dehydrogenase in a Japanese population."
T. Takeshita et al. Hum Genet (1994) 94:217-223
この論文ではPCRにより人工的な制限酵素サイトを導入しています。つまりReverseプライマーのTTCAC部位をTTCTCに変えて、そのPCR産物を制限酵素で処理することによりALDH2の遺伝子型を決定しています。そのため、このReverseプライマー配列と、それに対応するALDH2のゲノム配列を比較すると、一塩基相補的でない部位があることが分かります(下記Tの部分)。
ALDH2ゲノム |
5'-GAA GTG AAA ACT GTG AGT GTG G-3' |
Reverse プライマー(野生型) |
3'-CTT CTC TTT TGA CAC TCA CAC C-5' |
弊社では、制限酵素処理をせずにPCRのみで遺伝子型を決定するために、まず標的部位が確実に増えている上記論文のプライマー配列を参考にしました。そのため、上記論文で制限酵素サイトを導入するために入れたミスマッチ部位はそのまま残してあります。(補足:ミスマッチ部位を訂正したプライマーを使用した際、マニュアルのPCR条件下で非特異的増幅が認められたことがあります。)
補足情報.2 増幅領域について
上記のプライマーはALDH2のexon 12の全体を増幅します。NCBIのAccession No.はAH002599です。下記は2023.07.27現在のexon 12の情報です。最新の情報はNCBIのデータベースでご確認ください。塩基配列中のプライマー部位は赤文字で示してあります。
| LOCUS | AH002599 4002 bp DNA linear PRI 10-JUN-2016 | |
| DEFINITION | Homo sapiens aldehyde dehydrogenase (ALDH2) gene, complete cds. | |
| (中略) | ||
exon |
3325..3439 | |
| /gene="ALDH2" | ||
| /note="G00-119-668" | ||
| /number=12 | ||
| (中略) | ||
| ORIGIN | 3 bp upstream of HincII site; chromosome 12q24.2. | |
3315 |
caaatt acagggtcaa ctgctatgat gtgtttggag cccagtcacc | |
3361 |
ctttggtggc tacaagatgt cggggagtgg ccgggagttg ggcgagtacg ggctgcaggc | |
3421 |
atacactgaa gtgaaaactg tgagtgtgg | |
| // |
補足情報.3 ALDH2遺伝子について
ALDH2遺伝子は第12染色体長腕 (12q24.2) に位置し、44kbの塩基配列中に517個のアミノ酸をコードする少なくとも13個のエクソンを有しています。プロセシングを受けてシグナルペプチドが取り除かれるため、ALDH2の成熟タンパク質は500個のアミノ酸からなります。 ALDH2活性の欠損は主にALDH2遺伝子の点突然変異によるもので、ALDH2遺伝子の第12エクソンの114番目の塩基がG(グアニン)からA(アデニン)へ変異をおこしており、その結果、成熟タンパク質中の第487番目のアミノ酸がグルタミン酸(GAA)からリジン(AAA)に置換されています。
▲このページのトップへ
プライマー合成
ニッポンジーンでは、オリゴヌクレオチド合成サービスを承っております。ISOHAIR Jr.にプライマーは含まれておりませんので、スモールスケールオリゴ合成サービスやスタンダードオリゴ合成サービスをご利用いただくことで、ご希望のプライマーをカスタム合成することができます。本サービスのご利用方法はこちらをご覧ください。
オリゴヌクレオチド合成サービス
ALDH2プライマーセットをスモールスケールオリゴ合成サービスで注文する場合の注文方法
- ALDH2遺伝子の遺伝子型決定に使用する3本のプライマー(ALDH2プライマーセット)をニッポンジーンのスモールスケールオリゴ合成サービスでご注文された場合、下記の製品仕様でカスタム合成してお客様に提供されます。
-
<製品仕様> プライマー名 塩基配列 鎖長 保証収量*1 ALDH2 Forward CAAATTACAGGGTCAACTGCT 21 mer1 O.D.ALDH2 Reverse-N(野生型) CCACACTCACAGTTTTCTCTTC 22 mer1 O.D.ALDH2 Reverse-M(変異型) CCACACTCACAGTTTTCTCTTT 22 mer1 O.D.<スモールスケールオリゴヌクレオチド> 希望納入価格*25,650円(3本セット、精製込)合成スケール0.02 μmol(スモールスケール合成)精製*3カートリッジ精製納品形態100 μM水溶液納期*42営業日
- *1 保証収量として、1 O.D. 以上のオリゴヌクレオチドのご提供を保証していますが、容量のご指定はできません。容量の目安としまして、例えば22 merのオリゴヌクレオチドの場合、1 O.D. は約4.5 nmol (45 μl の100 μM 水溶液)に相当します(目安:一人当たり2サンプルで4反応すると想定した場合、40人分の実験に使用できます)。 100 μM水溶液を5倍希釈すると20 pmol/μL水溶液に調製できます。
- *2 希望納入価格に消費税は含まれておりません。
<参考: 3本セットの希望納入価格 の内訳> 5,650円 = Forward primer 1本(21 mer x 50円 + 800円 = 1,850円) + Reverse-N primer 1本(22 mer x 50円 + 800円 = 1,900円) + Reverse-M primer 1本(22 mer x 50円 + 800円 = 1,900円) - *3 PCR実験の場合、精製は脱塩グレードで十分な場合が多くありますが、ALDH2遺伝子型実験ではリバースプライマーの一塩基の違いを見ているため、カートリッジ精製を選択しています。
- *4 納期は、受注日を「0」とした場合の出荷日です。
<ご注文方法>
以下の申込用紙(Excel)をダウンロードして、必要事項をご記入後、内容を確認してニッポンジーンまでお送りください。
スモールスケールオリゴ申込用紙(配列入力済み) エクセルファイル
オリゴ合成サービスのご注文の流れ
https://nippongene-oligo.com/support/order-flow/
▲このページのトップへ
Q & A
- 実験例のプライマー終濃度は?
- 実験例では、20 μM(20 pmol/μL)濃度のプライマー溶液を50 μL反応系に1 μLずつ添加しているため、終濃度は0.8 μM each になります。使用されるPCR機器等に応じて、プライマー終濃度は 0.2 - 1 μMの範囲で最適なPCR条件をご検討いただくことをお勧めします。
- 増幅しない。
- 鋳型DNA量が少なすぎる可能性があります。ISOHAIR Jr.マニュアルに記載のDNA抽出のトラブルシューティングをご参考ください。
また、PCR用試薬を変えてみることも有効です。例えば、Gene Taq NT の代わりに Gene RED PCR Mix Plus を使用するなどの方法があります。 - 実習用キットISOHAIR Jr. 実験例より、もっと操作を簡略化させることはできますか?
-
実習の目的にもよりますが、プレミックスタイプや簡易プロトコールの試薬に置き換えると可能になります。例えば、ISOHAIR の代わりにISOHAIR EASY もしくはTemplate Prepper for DNAを使うとフェノール抽出の工程が省略され、Gene Taq NT の代わりに Gene RED PCR Mix Plus を使用すると反応液調製の時間が簡略され、Marker 4, 5の代わりに チューブからそのままアガロースゲルにアプライすることができる OneSTEP Marker 4 やGene Ladder 100を使用するなどの方法があります。
※試薬を変更する場合、必ずPCR反応系に添加するDNA量などについて事前検討してください。
Gene RED PCR Mix Plus を使用したALDH2遺伝子型決定のPCR実験例 (PDF 174KB)
▲このページのトップへ
関連情報
備考
- 本品は試験研究用試薬です。医薬品の用途には使用しないでください。
- ISOHAIR Jr.を用いて、人(試料・情報を含む。)を対象として実験を行う場合には、個人情報の保護に配慮して下さい。参考:「人を対象とする医学系研究に関する倫理指針ガイダンス」(文部科学省様サイト)
関連製品
- ISOHAIR (毛髪・爪からのDNA抽出キット)
- ISOHAIR Jr. (ISOHAIR, PCR用試薬, アガロースゲル電気泳動用試薬のセット)
- Gene Taq NT(PCR用試薬)
- Agarose 21(アガロース)
問い合わせ先
- 購入に関するお問い合わせ先
- 富士フイルム和光純薬株式会社および同社代理店・特約店
- 富士フイルム和光純薬株式会社 製品検索サイト
- 製品に関するお問い合わせ先
- 株式会社ニッポンジーン 学術営業課
